12 Aug
应用分享 | Dragonfly的空间转录原位测序和多重成像应用
研究挑战
对生物发育、脑功能、神经退行性疾病以及癌症的分子机制和行为的了解是一项繁重的工作。直到最近,大容量以及单细胞水平的RNA测序推进了科学家对上述问题的进一步了解,但遗憾的是对于组织环境相关信息了解的比较少。单分子荧光原位杂交(smFISH)技术帮助科学家获得RNA的空间信息,但是也仅仅针对少量的可以区分的基因才能实现。
获取2D(最好是3D)组织环境的信息能够帮助建立基因表达网络,从而揭示组织功能和相关疾病的潜在机制。现代细胞与分子生物学的圣杯是通过对同一块样品上的多个(Xn)RNA信息的绘制探究整个样品中的基因表达网络。
细胞生物学中的多重检测指的是在2D或者3D的层面对多个(Xn)RNA(或者其他生物分子)进行检测。多重检测是目前科研中的研究热点,在神经科学,肿瘤生物学,疾病靶向诊断,发育及行为学分析等科研中都发挥着重要的作用。多重检测能够获取多个基因产物的空间和序列信息,由此新技术发展起来的会产生重大影响的应用数量将会持续快速的增加。
在RNA生物学中,已经有许多发展起来的技术能够实现多重检测。每种技术都有其优点和缺点,但都有相同的目标,即建立基因表达的空间(视觉)图谱,及其与细胞和组织背景的关联性。类似的技术包括:FISSEQ(2)、instaSEQ(3)、osmFISH(4)、STARmap(5)、MERFISH(6)和seqFISH(7)。这些方法在科学界的相关性正在不断增强。其重要性的一个例子是,空间解析转录组学被Nature选为2020年度方法。
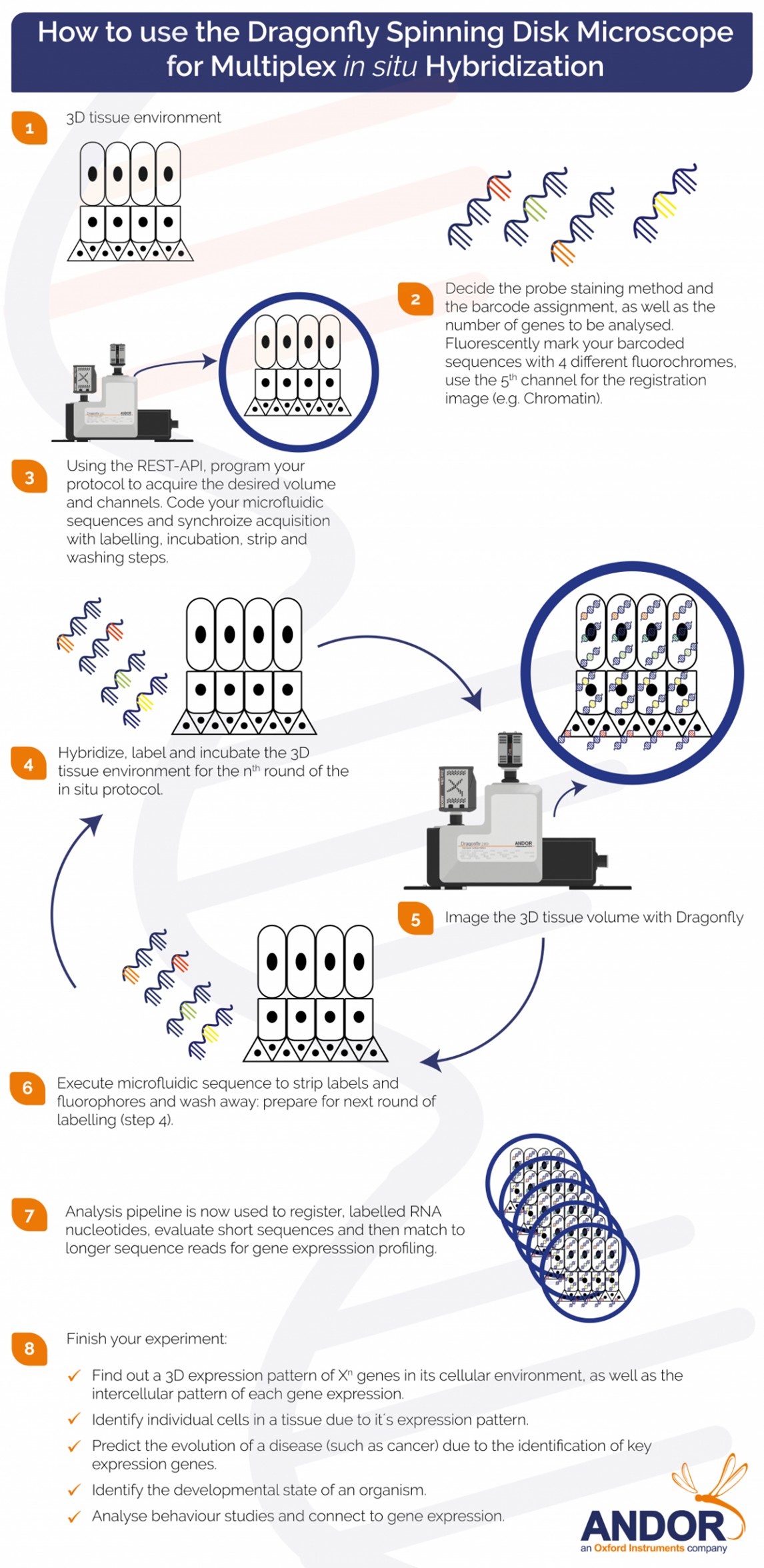
为了识别细胞(组织)内的基因表达量,必须在同一个样品上依次进行几轮杂交及检测。这种连续样品处理和图像采集过程一般是在搭载了微流控的显微成像系统上完成的。工作流程一般为:在杂交RNA分子上标记荧光探针,显微镜获取三维图像数据,然后洗去荧光探针;完成每一个图像数据的采集之后,开始下一个清洗-杂交轮次。经过多次的重复后,获取样品的大量待分析图像数据。图1为典型的图像数据集,展示了经过6个轮次的STARmap流程后获取的图像数据。图像中的每一个不同颜色荧光点代表着RNA序列中的每一个核苷酸;紧接着多重标记和图像采集之后的是离线的数据分析流程,这个流程将图像数据转化为这一次实验中所有靶向基因的表达图谱1。
在这个流程中,为了完成多重检测实验,需要克服几个挑战:比较基础的技术问题包括自动程序、采集速率、灵敏度和分辨率、光漂白、视场均匀性和数据通量;图像数据采集之后,有几个生物信息学的挑战需要考虑。无论是否存在偏移和非线性形状变化,每个视野(三维空间)内的数据必须是空间上匹配的,以保证按照顺序标记的核苷酸的准确测序并且明确分配到某一基因产物或者mRNA分子。
技术方案
基于二维成像相机的多点共聚焦系统非常适用于多重原位成像。此类成像系统要求具有较低的背景噪音,较高的灵敏度及分辨率以获取目标基因产物的精确定位。由于实验程序的复杂及持续时间长等特性,采集速度是获取更高产出数据量的重要参数。所以,能够同时获取多个波长的数据也是锦上添花的特征参数。
样品的相同区域需要多次重复成像,因此程序的每一轮次都要求系统具有高度稳定性。系统必须在多个轮次的成像及连续标记期间保持聚焦稳定性,在此期间微流控的温度和条件有可能发生了某些变化。
成像系统必须能够提供温和的成像条件,在尽量低的光漂白的情况下获取数据。为达成此项条件,探测器必须满足高灵敏度和高动态范围,不仅仅需要能够采集弱光信号,还需要捕捉样品发出的所有信号。当细胞核的DNA信号也作为采集程序的一部分时,信号可能在几个数量级之间变化。因此,高动态范围是探测器必须具备的参数。
对几个平方毫米范围的组织进行的三维成像时,需要在较高的成像深度上获取高分辨成像数据,因此理想的成像系统所有激发光在成像视野的照明必须是高度均匀的,保证多视野拼接获得的数据集的成像质量,在整个成像范围内保持高信噪比、有助于靶向产物的精确检测、识别及成像绘制,为下一步的分析提供保证。
单轮次的多重原位成像过程可能会耗费几个小时,6-10个轮次的整个流程花费的时间可能超过24小时。因此,软件、硬件和微流控系统可以整合并同步执行其各种任务是非常重要的。这种集成必须是满足操作灵活并且按照实验需求自主调节实验流程。
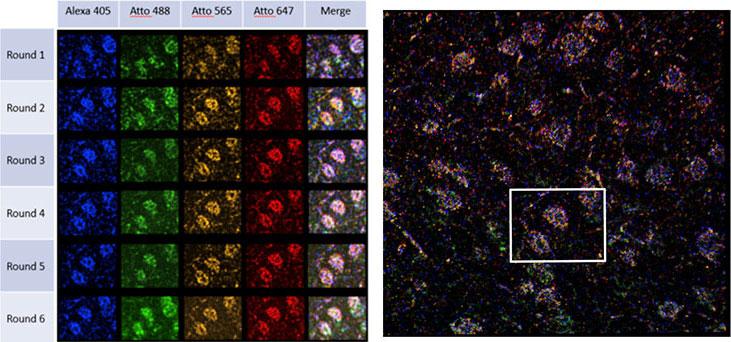
图1-使用STARmap对小鼠海马连续标记,由Dragonfly成像。左图为展示的20微米厚度的单个视野MIP图,拍摄调节为4个通道,71层(0.3um单层);右图展示的是拼接而成的大视野图像。资料来源于纽约哥伦比亚大学基因组学中心的Abbas Rizvi博士。
ANDOR多重原位杂交成像解决方案
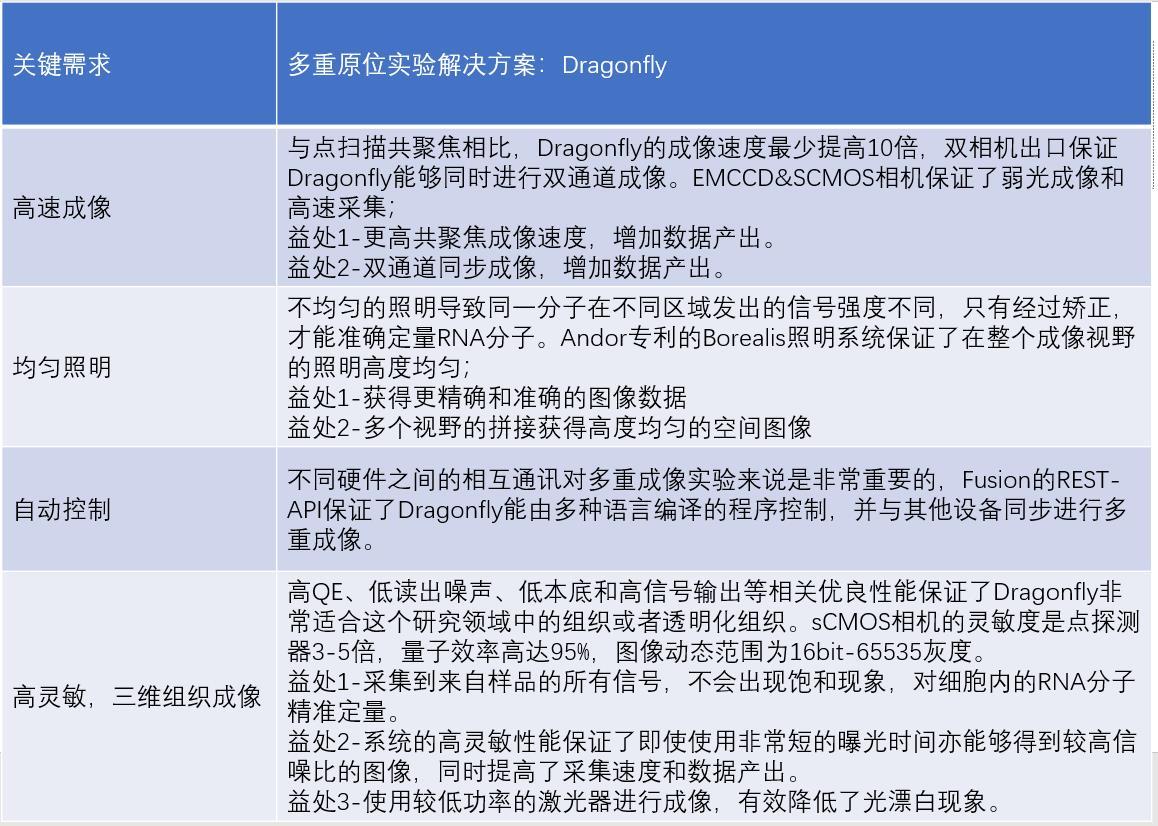
Dragonfly高速共聚焦成像系统非常适合原位杂交成像。从硬件上来说,Dragonfly属于多点扫描共聚焦系统,为科研工作提供高质量的共聚焦图像,同时具有非常高的成像速度(最高400fps);同样重要的是,Dragonfly也具有较大的成像视野(FN22),这保证了对较大块状样品的快速采集;此外,双微透镜转盘系统与Andor高动态范围和高QE相机(如背照式Sona sCMOS和iXon EMCCD系列相机)保证了整个系统能够采集到来自于样品的所有信号(从最弱到最强的信号)。
采集多重原位实验的数据时,对于视场照明的均匀性和数据通量有很大要求。Andor的Borealis照明系统保证了Dragonfly在整个成像视野中具有优良的均匀性,同时对多个视场和多个波长进行成像,有效的提高了数据通量。
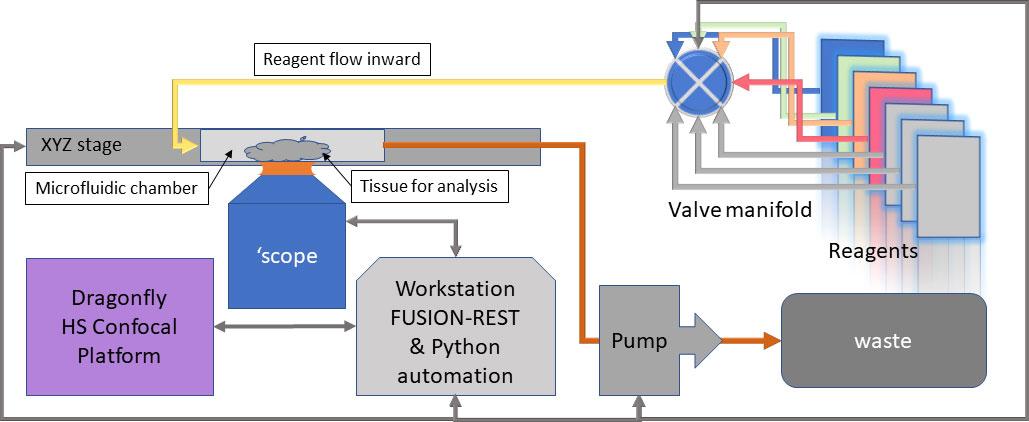
最近Andor将REST-API功能耦合进Fusion软件,允许从外部程序(如Python, LabView, MatLab)控制Dragonfly。这样就在Dragonfly与微流控系统间建立了通讯关系,用于杂交、标记、清洗相关试剂的连续流入;Dragonfly控制硬件用于图像采集;此图像采集功能可以按需触发以保证同步获取多通道三维数据。研究人员一旦熟悉编译环境,更容易控制微流控和成像程序的改变,图2展示了整个操作流程。
Dragonfly具有高速、高分辨成像能力,与大视野成像、优良的本底抑制以及Borealis均匀照明系统的能力使Dragonfly成为研究原位测序(转录组学)的强大工具。这些功能与REST API结合后为科研工作者提供高度灵活的多重成像系统。
科学家将反复探测、成像和去染色,以仅从一张组织图像中捕获数千个空间细胞数据点GE Cell Dive 多重成像。
注释
1. Multiplexing increases identifiable gene products (GP) rapidly. With 4 labels per round, R the number of GP’s which can be labelled uniquely is 4R. Theoretically, nine rounds could label all gene products in the human genome!
参考文献
- Raj A, Peskin CS, Tranchina D, Vargas DY, Tyagi S. Stochastic mRNA synthesis in mammalian cells. PLoS Biol.
2006;4(10):e309. doi:10.1371/journal.pbio.0040309 - Lee JH, Daugharthy ER, Scheiman J, Reza Kalhor R., Ferrante T.C., Terry R., Turczyk B. M., Yang J.L., Lee H. S., Aach
J., Zhang K. & Church G. M. Fluorescent in situ sequencing (FISSEQ) of RNA for gene expression profiling in intact cells and tissues. Nat Protoc. 2015;10(3):442–458. doi:10.1038/nprot.2014.191 - Daniel Fürth, Victor Hatini, Je H. Lee. In Situ Transcriptome Accessibility Sequencing (INSTA-seq) bioRxiv 2019; 722819; doi:https://doi.org/10.1101/722819
- Codeluppi S, Borm LE, Zeisel A, La Manno G., van Lunteren J. A., Svensson C. I.& Linnarsson S. Spatial organization of the somatosensory cortex revealed by osmFISH. Nat Methods. 2018;15(11):932–935. doi:10.1038/s41592-018-0175-z
- Wang X, Allen WE, Wright MA, Sylwestrak E. L., Samusik N., Vesuna S., Evans K., Liu C., Ramakrishnan C., Liu J., Nolan G. P., Bava F. & Deisseroth K. Three-dimensional intact-tissue sequencing of single-cell transcriptional states. Science. 2018;361(6400):eaat5691. doi:10.1126/science.aat5691
- Chen KH, Boettiger AN, Moffitt JR, Wang S, Zhuang X. RNA imaging. Spatially resolved, highly multiplexed RNA profiling in single cells. Science. 2015;348(6233):aaa6090. doi:10.1126/science.aaa6090
- Shah S, Lubeck E, Zhou W, Cai L. In Situ Transcription Profiling of Single Cells Reveals Spatial Organization of Cells in the Mouse Hippocampus. Neuron. 2016;92(2):342–357. doi:10.1016/j.neuron.2016.10.001



 公安机关备案号31010402003473
公安机关备案号31010402003473